L'IA au service de la résolution de la longue énigme du COVID
11 février 2022
11 février 2022
Par Jonathan Woods
Cette affection porte plusieurs noms : syndrome post-COVID, COVID longet séquelles post-aiguës du SRAS-CoV-2. C'est l'écho angoissant de COVID-19, et nombreux sont ceux qui ont eu la malchance d'en faire l'expérience. Plus de la moitié des personnes ayant contracté le COVID-19 ont signalé au moins un symptôme persistant trois mois après l'infection. Selon l'Agence de santé publique du Canada, les symptômes courants de COVID à long terme comprennent la fatigue, l'essoufflement et les troubles de la mémoire, bien que "plus de 100 symptômes ou difficultés dans les activités quotidiennes" aient été signalés. Pour certains, la maladie est débilitante et semble indéfinie[1]. [1]
La COVID longue n'est pas bien comprise. "Nous ne connaissons pas les causes de l'état post-Covid-19", peut-on lire sur le site web de l'agence de santé publique. "Il n'existe actuellement aucun moyen de diagnostiquer l'état post-Covid-19 et il n'y a pas de traitement à l'heure actuelle. [1]
L'IA peut-elle contribuer à accélérer la découverte des réponses ?
Les équipes Innovation industrielle et Ingénierie de l'IA de l'Institut Vecteur ont travaillé avec Roche Canada, Deloitte et TELUS pour explorer cette question. Leur projet : appliquer des techniques de traitement du langage naturel (NLP) aux messages publiés sur les médias sociaux par des personnes atteintes d'une COVID de longue durée afin de voir si des schémas se dégagent. Il s'agissait de créer un pipeline d'apprentissage automatique et de tester les capacités de divers modèles de traitement du langage naturel sur des témoignages de première main tirés des médias sociaux. L'espoir est que ces schémas, s'ils sont identifiés, puissent révéler des indices sur le moment et la fréquence d'apparition des symptômes, ainsi que sur les endroits où l'on trouve des groupes de personnes atteintes de la maladie. Ces informations pourraient être partagées avec les cliniciens afin d'affiner leurs questions de recherche, d'identifier les tendances à un stade précoce ou d'éclairer les stratégies de traitement.
"Les patients se tournent souvent vers les médias sociaux pour exprimer leur expérience de la maladie", peut-on lire dans le rapport sur l'utilisation des médias sociaux dans le secteur de la santé. L'utilisation des médias sociaux dans les soins de santé, un document de recherche sur l'utilisation des réseaux sociaux par les patients. Le soutien informationnel, émotionnel et d'estime sur les réseaux sociaux "conduit généralement à l'autonomisation du patient", poursuit le document[2]. Cela encourage le partage et peut faire des médias sociaux une ressource riche pour les chercheurs - mais seulement s'ils peuvent filtrer de manière fiable l'océan de messages publiés chaque jour et repérer ceux qui contiennent un langage pertinent.
Même pour les modèles d'apprentissage automatique avancés, il s'agit d'un défi.
Elham Dolatabadi, chercheur en apprentissage automatique appliqué à l'Institut Vecteur et responsable technique du projet, explique : "L'extraction d'entités médicales à partir des médias sociaux est un défi en raison de la nature non structurée du contenu, qui est souvent bruyant, informel et court. Sans compter que la complexité des termes médicaux entraîne parfois des fautes d'orthographe." Les messages générés par les utilisateurs sont loin d'être uniformes, et leur brièveté et leur manque de structure (qui inclut l'utilisation de l'argot et les différences de ton) rendent difficiles l'identification, l'extraction et la classification des récits de première main de COVID longs.
Pour relever ces défis, l'équipe a développé un pipeline d'apprentissage machine (ML) personnalisé spécialement conçu pour trouver, ordonner et afficher les longs termes COVID qui seraient autrement enfouis dans une montagne de messages sur les médias sociaux. Le pipeline organise le processus de bout en bout, regroupant la collecte et le filtrage des données, l'entraînement de divers modèles pour extraire et classer les termes clés des messages, et la visualisation des résultats sur des tableaux de bord en un seul processus taillé sur mesure pour cette tâche.
La première étape du développement du pipeline a consisté à créer des ensembles de données constitués de messages provenant de Twitter et de Reddit. À l'aide de l'interface de programmation d'applications (API) de Twitter, l'équipe a recherché des tweets contenant des hashtags pertinents - comme #longcovid, #postcovidsyndromeet #covidlonghauler - ainsi que des termes similaires non marqués d'un hashtag. Une fois collectées, les données ont été passées au crible d'un filtre de type "longhauler" conçu pour identifier les témoignages à la première personne. Ce processus a également consisté à supprimer les messages comportant des titres d'actualité ou des noms de produits exclusifs. Les tweets restants ont été dépersonnalisés, toutes les informations personnelles concernant l'auteur du message ayant été supprimées (les horodatages, les informations géographiques et les descriptions générales du profil de l'utilisateur ont toutefois été conservés).
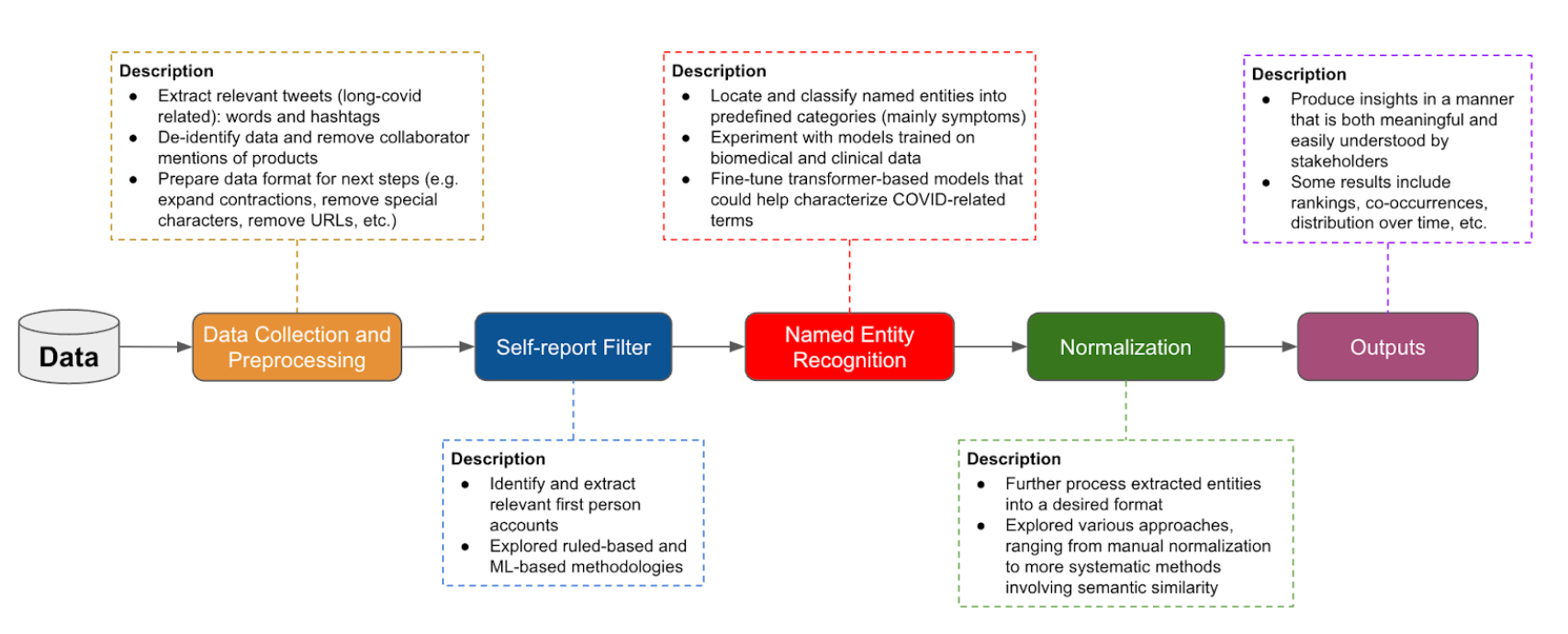 Figure 1. L'équipe a construit un pipeline de ML personnalisé pour collecter, filtrer, analyser et visualiser les longues informations COVID glanées dans les messages des médias sociaux.
Figure 1. L'équipe a construit un pipeline de ML personnalisé pour collecter, filtrer, analyser et visualiser les longues informations COVID glanées dans les messages des médias sociaux.
Avec les ensembles de données en main, l'équipe a testé un ensemble de modèles spécialisés formés à une technique de NLP appelée reconnaissance des entités nommées (NER). La NER identifie des entités spécifiques (comme des personnes, des lieux et des objets) dans un texte et les classe ensuite dans des catégories prédéfinies. Ces expériences ont été conçues pour voir si les modèles, formés spécifiquement pour extraire des termes médicaux, pouvaient identifier le langage dénotant un symptôme, un test ou un traitement COVID long, puis le classer avec précision en tant que tel. La figure 2 illustre le fonctionnement de ces expériences sur deux exemples de tweets.
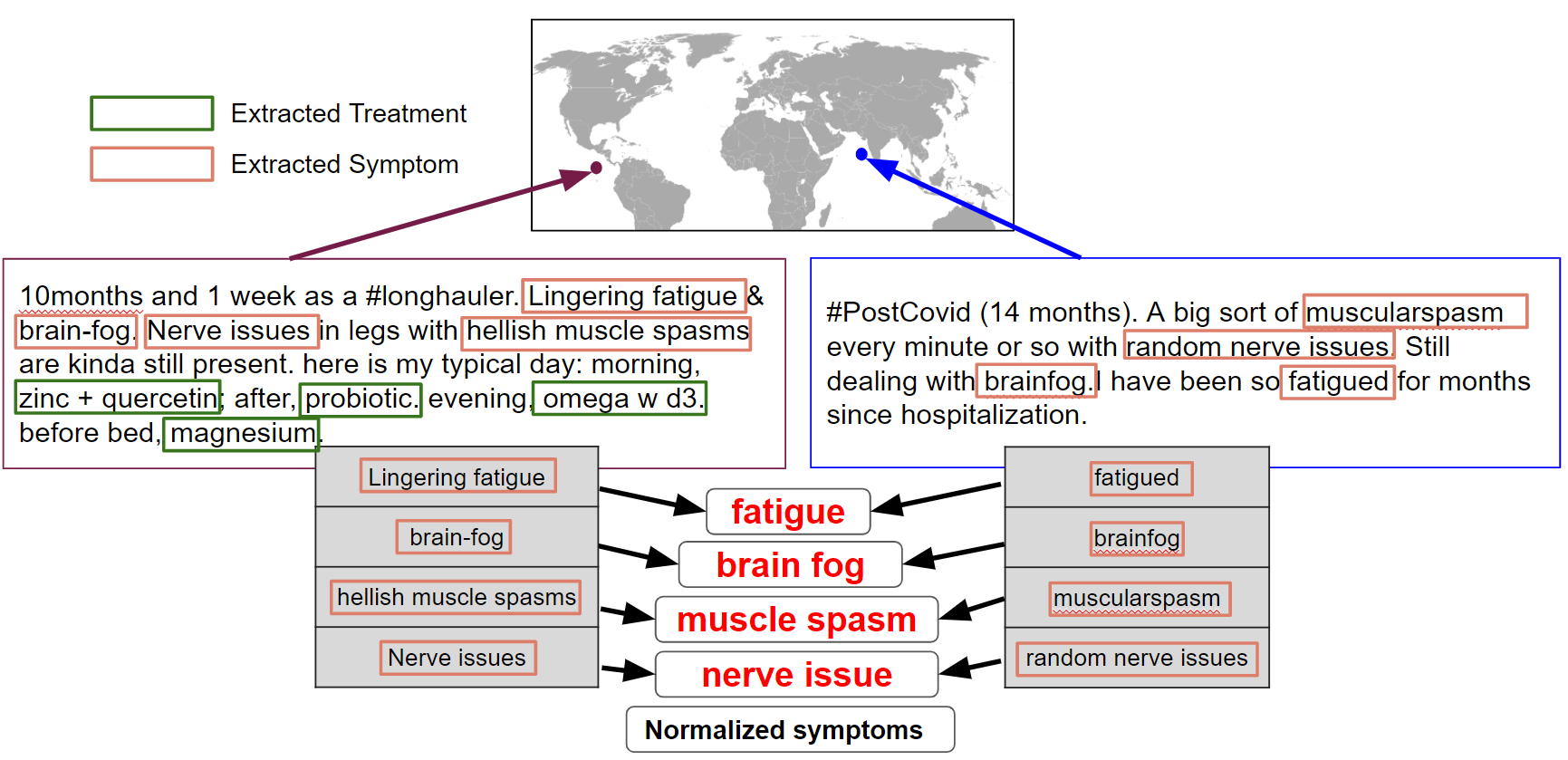
Figure 2. Parmi les modèles étudiés par l'équipe figure Stanza, un modèle de NER créé par Stanford. Stanza a été entraîné sur un corpus de textes biomédicaux et cliniques et est utilisé pour identifier les symptômes ou les traitements dans les messages des médias sociaux, et pour les extraire, les classer et les normaliser de manière appropriée. Notez que les exemples sont créés synthétiquement.
L'un des modèles testés par l'équipe était UmlsBERT, un modèle déjà entraîné sur un large corpus de métathésaurus cliniques (UMLS Metathesaurus). Une partie de l'expérience consistait à affiner ce modèle sur un ensemble de données cliniques fournies par le National Center for Biomedical Computing (NCBC), connu sous le nom d'i2b2, et à augmenter les données pour permettre une extraction d'entités plus granulaire. En utilisant UMLS' MetaMapLite et l'ensemble de données AMIA Task3, l'équipe a amélioré l'extraction d'entités afin d'être en mesure de repérer même les termes familiers et informels, tels que "brouillard cérébral" et "fatigue écrasante".
Après avoir mené une série d'expériences, les résultats préliminaires ont montré qu'il était possible de détecter et de visualiser des modèles liés à la fréquence des symptômes, à leur cooccurrence et à leur distribution dans le temps. Les chercheurs peuvent en effet trouver des signaux dans le bruit des médias sociaux.
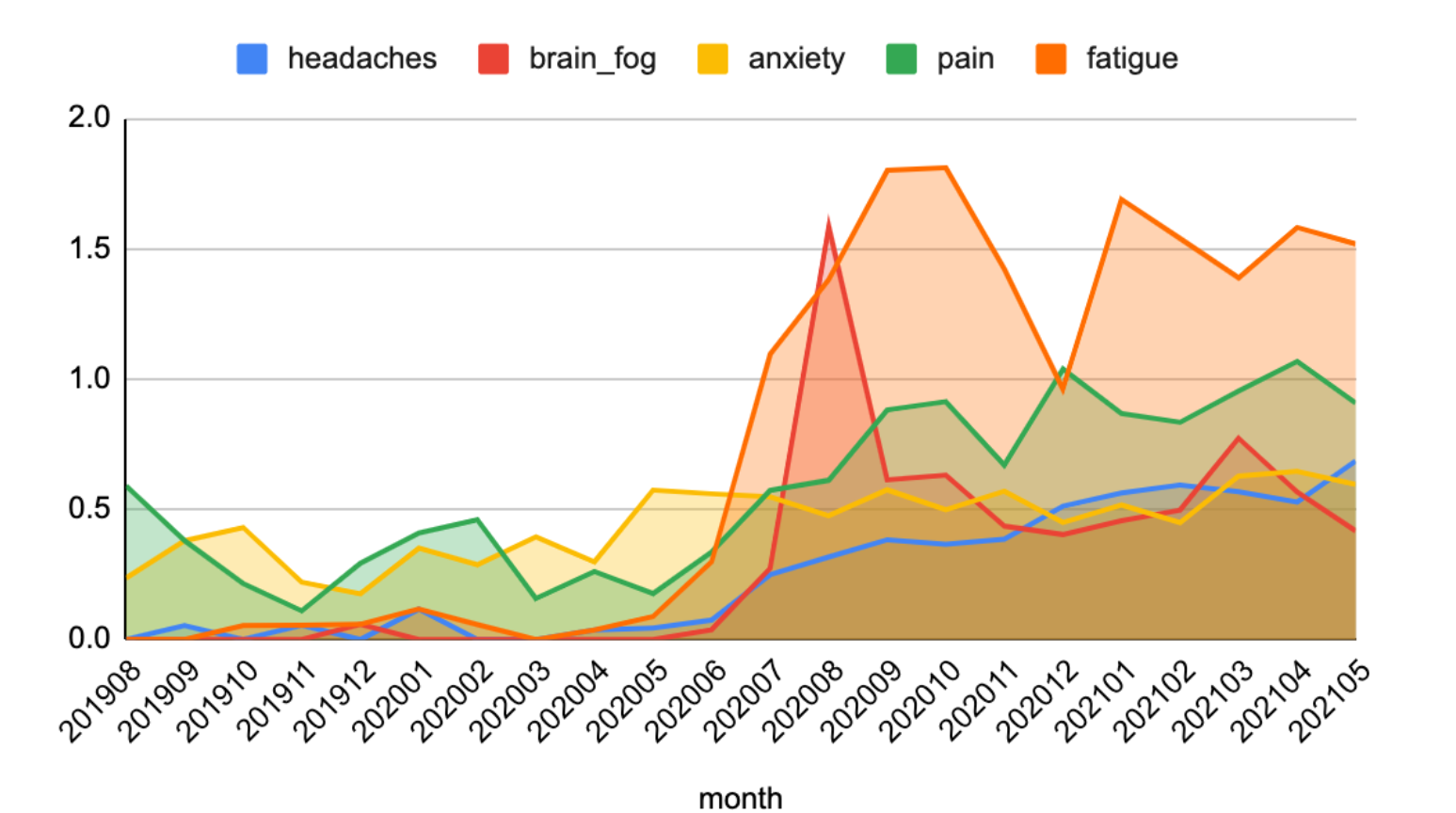
Figure 3. Fréquence d'apparition des cinq (5) principaux symptômes au fil du temps à l'aide du modèle Stanza et des données Twitter avec un filtre COVID spécifique et une normalisation.
Avec la confirmation de l'efficacité du pipeline pour l'extraction d'entités liées au COVID à partir des médias sociaux, la prochaine étape consiste à travailler avec les chercheurs et les cliniciens pour interpréter ces modèles et déterminer les orientations de recherche prometteuses qu'ils peuvent révéler.
"Nous pouvons utiliser cette approche pour donner aux cliniciens plus d'informations et fournir des signaux précoces qui peuvent les aider à planifier leurs études cliniques, leurs traitements, leurs thérapies, etc.", déclare Sedef Akinli Kocak, chef de projet chez Vector. "C'est un excellent exemple de collaboration entre les chercheurs, les experts en la matière et les entreprises qui souhaitent contribuer à la lutte contre le COVID 19".
Les entreprises participantes, les experts en matière médicale et les contributions des doctorants de diverses institutions canadiennes méritent une mention spéciale. Ensemble, tout au long du projet, Roche Canada, Deloitte, TELUS et les chercheurs au doctorat ont fourni l'expertise clinique et d'apprentissage automatique qui a permis au pipeline de fonctionner. Les principales contributions de ces entreprises commanditaires du vecteur et des chercheurs au doctorat comprennent l'idéation originale du projet, l'examen de la documentation clinique sur les longs courriers COVID, la collecte, le nettoyage et l'annotation des données, la mise en œuvre des filtres, la normalisation, la modélisation NER, l'ingénierie de la visualisation et l'interprétation des résultats.
La valeur de ces contributions ira au-delà de cette pandémie. Les chercheurs disposent désormais d'un outil et d'un processus permettant de faire des médias sociaux une ressource clé pour comprendre d'autres événements sanitaires au niveau de la population, tels que les nouvelles maladies infectieuses émergentes, les maladies rares ou les effets des vaccins de rappel sur l'infection dans diverses régions et à divers moments.
Pour l'instant, l'accent reste mis sur les personnes souffrant de COVID longue. On espère que cette nouvelle capacité permettra aux chercheurs et aux cliniciens de faire un pas de plus vers la résolution des mystères de la COVID longue et de jouer un rôle dans l'allègement des souffrances de ceux qui en ressentent les effets.
Un document technique lié au projet a été accepté pour être présenté à la conférence de l'Union européenne. 6e atelier international sur l'intelligence sanitaire lors de la conférence AAAI 2022. Un rapport complet sur les résultats du projet sera publié au début de l'année 2022. Pour l'instant, le projet est en cours.
Références :
[2] Smailhodzic, Edin, et al. "Social media use in healthcare : a systematic review of effects on patients and on their relationship with healthcare professionals." BMC health services research 16.1 (2016) : 1-14. LIEN : https://


